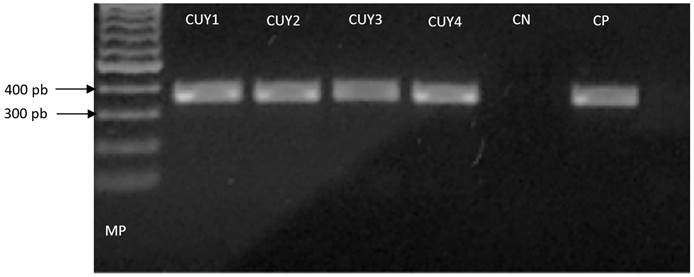
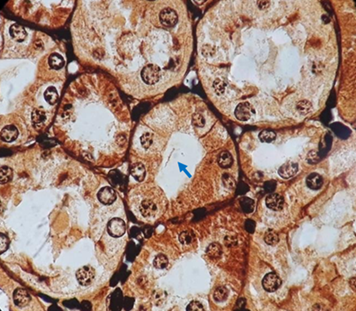
Este estudio es el primer reporte de infección natural por
Leptospira
interrogans en cuyes (Cavia porcellus) en el municipio de
Pasto. Se
logró detectar la presencia de Leptospira en muestras de riñón
mediante
dos métodos de detección totalmente diferentes (PCR convencional e
histopatología), por lo cual se corrobora la infección natural de
Cavia
porcellus. De 270 muestras de tejido renal analizadas, cuatro
fueron
confirmadas por PCR y una por PCR y tinción W-S. Se debe destacar que la
tinción W-S fue menos eficiente en detectar la presencia de
Leptospira
en riñones de Cavia porcellus que la PCR convencional. Estos
resultados
son congruentes con diversas investigaciones en las cuales se resalta
que la PCR tiene una mayor sensibilidad en el diagnóstico de la
Leptospirosis que las pruebas histopatológicas21. Sin embargo, se sugiere que la mejor
herramienta para el diagnóstico de la leptospirosis animal es realmente
una combinación entre las pruebas serológicas, moleculares y tinciones
histopatológicas, debido a que dicha combinación aumenta la sensibilidad
y especificidad del diagnóstico.
Adicionalmente, esta combinación brinda a los investigadores la
posibilidad de detectar un mayor número de animales infectados, la
capacidad de medir el contacto previo de los animales con la bacteria
mediante pruebas serológicas, detectar la infección de animales a partir
de diferentes muestras de tejidos y fluidos mediante PCR y evaluar los
daños ocasionados por la bacteria en los diferentes órganos mediante
tinciones histopatológicas21.
Por lo tanto, los hallazgos de este trabajo se convierten en la base
para desarrollar estudios que permitan esclarecer el rol de la especie
Cavia porcellus en la epidemiología de la leptospirosis en el
sur de
Colombia.
La mayoría de los reportes sobre L. interrogans en cuyes son de
tipo
experimental, en los cuales esta especie se usa como biomodelo para
establecer rutas de entrada, lesiones causadas en órganos blanco
(hígado, riñón y pulmón) y evaluación de la respuesta inmunitaria
mediante la inoculación de la bacteria a diferentes dosis22,23. En
2020, un estudio realizado en granjas de cuyes familiares y comerciales
en Cajamarca-Perú, reportó una seroprevalencia de 40,50% de anticuerpos
contra Leptospira spp. mediante la prueba de migroaglutinación
(MAT),
identificando los serovares Icterohaemorrhagiae 19,01%,
Canicola 16,53%,
y Pomona 8,68%24. Otro
estudio identificó los factores asociados a seropositividad de
Leptospira spp. en humanos y reportó que la tenencia de cuyes
como
mascotas es un factor de riesgo para la infección25. Finalmente, en Colombia, un estudio
realizado en el departamento de Putumayo resaltó que la susceptibilidad
de los cuyes a Leptospira aumenta la posibilidad de transmisión
de la
enfermedad a los humanos y otras especies animales, y recomienda
desarrollar investigaciones complementarias para identificar la
importancia de la especie Cavia porcellus en la cadena
epidemiológica de
la enfermedad26.
Una revisión sistemática sobre la prevalencia de la leptospirosis en
Colombia utilizando pruebas de diagnóstico como Microaglutinación (MAT)
y Enzimoinmunoanálisis de adsorción (ELISA) indica variabilidad en las
diferentes poblaciones reportando que las prevalencias encontradas están
entre 6% y el 35% para humanos, 41% y 60,9% para bovinos, 10,3% para
porcinos, 12% y 47,14% para caninos, 23,07% para primates no humanos y
entre el 25% y el 82,7% en roedores, siendo las más altas27.
La alta prevalencia de roedores infectados juega un papel importante en
la trasmisión de Leptospira a otros mamíferos, incluyendo al
humano28. Por lo tanto,
debido a las
características en los sistemas familiares de producción de cuyes en
Nariño, en los que se resalta la ausencia de instalaciones de crianza;
no existen barreras físicas que impidan el ingreso de roedores
silvestres u otras especies de animales domésticos, permitiendo el
contacto directo con los cuyes y la potencial contaminación del agua y
comida con orina y heces, permitiendo la diseminación del agente en esta
población9. Adicionalmente,
las condiciones ambientales generadas por el manejo de los animales
(camas con material orgánico y alimentación en el suelo), crean
ambientes cálidos y húmedos, ideales para la supervivencia de la
bacteria28.
En los sistemas de crianza tanto familiares como comerciales no es
frecuente el uso de elementos de protección personal en el momento de
manipular los animales o el material orgánico en contacto con la orina,
facilitando la exposición de las personas a la bacteria3. De igual manera, durante el
proceso de sacrificio de los cuyes se observó la ausencia de cualquier
tipo de protección personal. La falta de uso de estos elementos podría
explicarse por el desconocimiento de los riesgos biológicos por parte de
la población y por la falta de control oficial en la aplicación de la
normatividad que regula el funcionamiento de plantas de sacrificio para
productos cárnicos provenientes de especies domésticas como el cuy29. En estudios realizados en
otras especies se identificó que el uso inadecuado o ausencia del equipo
de protección personal para los trabajadores de los mataderos es un
factor de riesgo para la exposición a Leptospira30,31.
Adicionalmente,
debe mencionarse que la identificación de Leptospira en plantas
de
sacrificio es un excelente método de vigilancia para la enfermedad
porque en la mayoría de los sistemas productivos es difícil
diagnosticarla dadas las características silenciosas de la infección32.
Por otro lado, hasta el momento no hay evidencia que sugiera que exista
riesgo de infección por el consumo de carne de animales infectados con
Leptospira. Al respecto, existen evidencias de la
desintegración de las
espiroquetas cuando son expuestas por períodos cortos de tiempo a
temperaturas superiores a 45 °C33,34, por lo cual el
proceso de cocción será suficiente para disminuir el riesgo. En el caso
de los cuyes, se pudo observar que dentro del proceso de sacrificio se
hace uso de agua caliente a una temperatura mayor a 60 °C para facilitar
el retiro del pelo, y posteriormente las vísceras son sometidas a
ebullición y asado sugiriendo que el riesgo para el consumidor final es
bajo. Sin embargo, para llegar a esta conclusión es importante
establecer cuáles son los factores de riesgo para la infección en esta
especie y en los humanos expuestos, así como las medidas de protección
personal que deben implementarse durante el manejo en los sistemas de
producción y los mataderos como se ha realizado con otras especies
animales25,30,35.
Finalmente, es necesario recordar que la leptospirosis en humanos es
mucho más frecuente de lo que se supone. Al respecto, la Organización
Mundial de la Salud (OMS) refiere que en algunos países esta enfermedad
no es correctamente diagnosticada y en otros, los casos no son
notificados como leptospirosis, debido a la similitud de sus signos y
síntomas con otras enfermedades como la malaria y el dengue36,37. En
Colombia, la leptospirosis hace parte de las zoonosis de notificación
obligatoria al Sistema Nacional de Vigilancia en Salud Pública
(Sivigila) desde el año 200738. Dentro de los informes de los casos
notificados al Sivigila en el período 2012-2016 se encontró que el 47,1%
de los casos tuvo contacto con animales domésticos como perros y el
46,9% con ratas en el domicilio36. En Nariño, durante el 2020 se reportó
un solo caso y a la semana epidemiológica 13 del año 2021 ya se han
reportado 11 casos39. La
deficiencia en la notificación de la enfermedad limita las actividades
de prevención y control en la población humana, reflejado en las
pérdidas socioeconómicas por las altas tasas de morbilidad y mortalidad,
obviando las situaciones de riesgo en la interfaz
humano-animal-ecosistema que convierten a la leptospirosis en una
zoonosis de alto impacto en la salud pública y de preocupación a nivel
mundial40.