Actividad antibacteriana de actinomicetos frente a Escherichia coli y Staphylococcus aureus multirresistentes de origen hospitalario
Antibacterial activity of actimycetes against multiresistant Escherichia coli and Staphylococcus aureus isolated from a hospital
Como citar: Ortega-Bastidas LA, Castillo-Arteaga RD, Gómez-Arrieta JD, Fernández-Izquierdo P, Burbano-Rosero EM. Actividad antibacteriana de actinomicetos frente a Escherichia coli y Staphylococcus aureus multirresistentes de origen hospitalario. Univ Salud. 2022; 24(1):85-94. DOI: https://doi.org/10.22267/rus.222401.257
Resumen
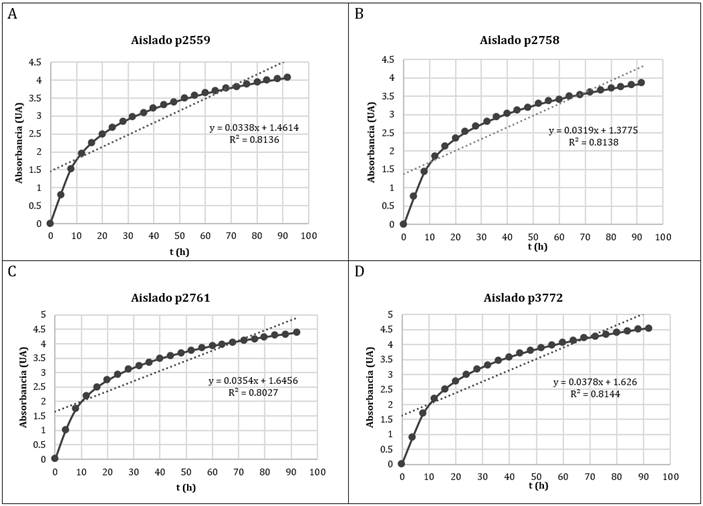
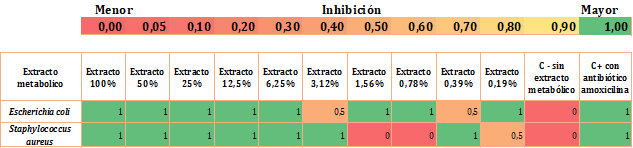
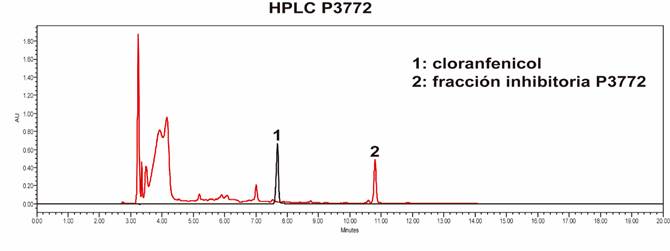
Introducción: La resistencia a los antimicrobianos (RAM) es un problema de salud pública que manifiesta la disminuida eficacia de estos agentes en la prevención y tratamiento de una proporción cada vez más amplia de patologías. Los actinomicetos son un grupo bacteriano importante de productores de metabolitos activos contra patógenos. Objetivo: Aislar actinomicetos del bosque tropical de Nariño, con potencial producción de metabolitos inhibitorios contra bacterias multidrogo-resistentes. Materiales y métodos: Se tomaron muestras de suelo de Bosque Tropical Húmedo de la Reserva Natural del Río Ñambí, se analizaron microbiológica y molecularmente. Se estimuló la producción in vitro de metabolitos secundarios y evaluó el efecto inhibitorio de estos extractos contra las bacterias multidrogo-resistentes Escherichia coli y Staphylococcus aureus. Resultados: Se obtuvieron 11 aislados presuntivos, se confirmó que cuatro de ellos correspondieron al género Streptomyces sp. Las pruebas de inhibición contra bacterias multidrogo-resistentes E. coli y S. aureus, permitieron verificar que el aislado P3772 fue el más eficiente en la inhibición de los patógenos. Conclusiones: Todos los actinomicetos evaluados presentan actividad antibacteriana contra al menos una de las bacterias patógenas estudiadas; destacando el aislado P3772, que inhibe a E. coli y S. aureus. Se espera caracterizar los compuestos vinculados a la actividad antibacteriana.
Palabras clave: Antibacterianos; farmacorresistencia bacteriana; Actinomycetales.
Abstract
Introduction: Antimicrobial resistance (AR) is a public health problem that reveals the diminished efficacy of these agents in the prevention and treatment of an increasingly larger number of pathologies. Actinomycetes are an important bacterial producer group of metabolites that are active against pathogens. Objective: To isolate actinomycetes from the tropical forest of Nariño (Colombia), which have the potential to produce inhibitory metabolites against multi-drug resistant bacteria. Materials and methods: Soil samples were taken from the Humid Tropical Forest of the Río Ńambí Natural Reserve and analyzed through microbiological and molecular assays. In vitro production of secondary metabolites was first stimulated, followed by the assessment of the inhibitory effect of these extracts against multi-drug resistant Escherichia coli and Staphylococcus aureus. Results: 11 presumptive isolates were obtained, confirming that four of them corresponded to the Streptomycess sp. genus. The bacterial isolate P3772 was identified as the one with the highest inhibitory effect against multi-drug resistant E. coli and S. aureus. Conclusions: All the actinomycetes evaluated presented antibacterial activity. The isolate P3772 stands out, which inhibited both E. coli and S. aureus. The compounds associated with this antibacterial activity will be characterized in future studies.
Keywords: Anti-bacterial agents; drug resistance, bacterial; Actinomycetales.